Vẽ biểu đồ ma trận đồ thị phân tán bởi R
Công nghệ - Ngày đăng : 08:14, 28/04/2016
2. Bạn gọi ứng dụng này vào chương trình R bằng lệnh:
> library(PerformanceAnalytics)
3. Đồng thời, bạn cho phép R nhận dữ liệu từ các định dạng bên ngoài bằng cách gõ dòng lệnh:
> library(foreign)
4. Nhập dữ liệu vào R
Ví dụ, dữ liệu ở đây được nhập từ file SPSS có tên là Vebieudo và bạn phải thiết lập cho R một đối tượng để nhận dữ liệu nhập vào. Ở đây đối tượng chọn có tên là Dulieu (bạn có thể đặt một tên bất kỳ). Bạn gõ lệnh
> Dulieu=read.spss("C:/Users/dovuxulo/Desktop/Vebieudo.sav",use.value.labels=TRUE,to.data.frame=TRUE)
Nếu nhận được thông báo như sau là bạn đã thực hiện đúng lệnh:
Warning message:
In read.spss("C:/Users/dovuxulo/Desktop/Vebieudo.sav", use.value.labels = TRUE, :
C:/Users/dovuxulo/Desktop/Vebieudo.sav: Unrecognized record type 7, subtype 18 encountered in system file
Lưu ý: Nếu không có dấu ngoặc kép hoặc các ký tự lạ trên bàn phím, bạn hãy chọn chúng trong mục Symbol của Microsoft Word rồi sao chép qua R bằng lệnh Copy và Paste.
5. Cho R phân tích đối tượng Dulieu, bạn gõ dòng lệnh:
> attach(Dulieu)
6. Kiểm tra nội dung dữ liệu, bạn gõ dòng lệnh:
> edit(Dulieu)
Khi đó, kết quả cho thấy, dữ liệu có 4 biến là: tuổi, huyết áp tâm thu, đường máu và cholesterol. Sau đó bạn đóng giao diện này lại.
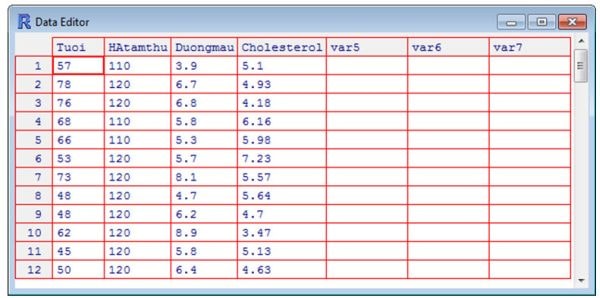
7. Kết hợp các biến vào một đối tượng có tên là mydata (bạn có thể đặt một tên bất kỳ), thông quia dòng lệnh:
> mydata=cbind(Tuoi,HAtamthu,Duongmau,Cholesterol)
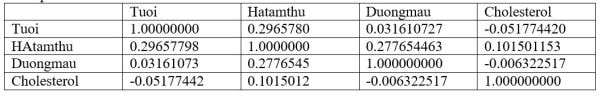
8. Xác định hệ số tương quan giữa các biến, bạn gõ dòng lệnh:
> cor(mydata)
Kết quả:
7. Bạn nhập đoạn lệnh sau vào R (lưu ý mỗi lệnh trên 1 dòng):
> cor.prob <- function (X, dfr = nrow(X) - 2) {
R <- cor(X, use="pairwise.complete.obs")
above <- row(R) < col(R)
r2 <- R[above]^2
Fstat <- r2 * dfr/(1 - r2)
R[above] <- 1 - pf(Fstat, 1, dfr)
R[row(R) == col(R)] <- NA
R
}
8. Sau đó, bạn nhập tiếp đoạn lệnh sau vào R:
> flattenSquareMatrix <- function(m) {
if( (class(m) != "matrix") | (nrow(m) != ncol(m))) stop("Must be a square matrix.")
if(!identical(rownames(m), colnames(m))) stop("Row and column names must be equal.")
ut <- upper.tri(m)
data.frame(i = rownames(m)[row(m)[ut]],
j = rownames(m)[col(m)[ut]],
cor=t(m)[ut],
p=m[ut])
}
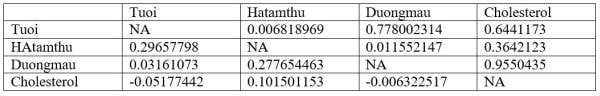
9. Xác định mức ý nghĩa p giữa các biến tương quan, bạn nhập dòng lệnh:
> cor.prob(mydata)
Kết quả:
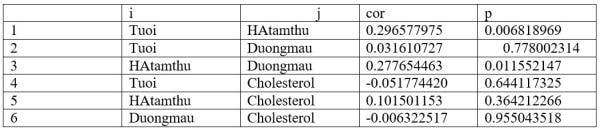
10. Trình bày hệ số tương quan và mức ý nghĩa p giữa các biến, bạn gõ dòng lệnh:
> flattenSquareMatrix(cor.prob(mydata))
Kết quả:
11. Vẽ biểu đồ ma trận đồ thị tương quan, bạn gõ dòng lệnh:
> chart.Correlation(mydata)
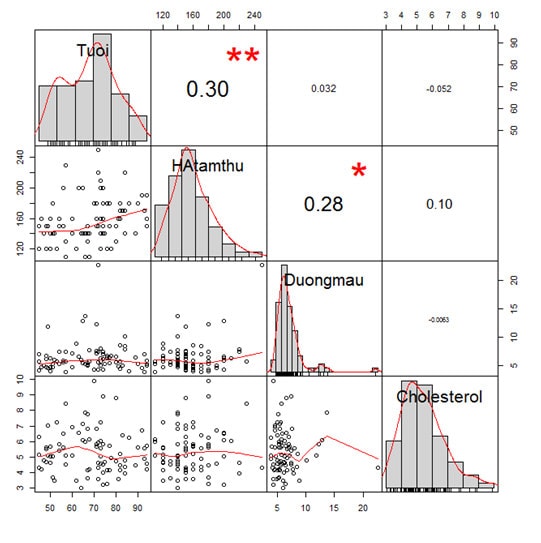
Biểu đồ cho ta rất nhiều thông tin như: phân phối dữ liệu của từng biến (chuẩn hay không chuẩn), hệ số tương quan r từng cặp biến, đồ thị phân tán của từng cặp biến, mức ý nghĩa thống kê p (cặp biến nào có dấu hoa thị là có ý nghĩa thống kê, nếu có hai dấu hoa thị trở lên là rất có ý nghĩa thống kê).
Lưu ý: R sử dụng kiểm định Pearson để tính hệ số tương quan. Kiểm định Pearson chỉ áp dụng cho 2 biến số có phân phối chuẩn. Vậy nên biến số nào không thuộc phân phối chuẩn thì ta phải log hóa biến trước để đạt phân phối chuẩn hoặc gần chuẩn, sau đó mới đưa vào biểu đồ ma trận.
Sau khi có được đồ thị, bạn hãy sao chép đồ thị vào bộ công cụ Office của Microsoft như Word, Excel, PowerPoint bằng cách nhấp chuột phải vào đồ thị rồi chọn Copy as metafile hoặc Copy as bitmap, sau đó thực hiện lệnh Paste; hoặc bạn có thể lưu đồ thị thành một tệp hình ảnh bằng cách chọn Save as metafile.
Bạn có thể tải dữ liệu tham khảo tại https://www.mediafire.com/?9v14yv718r8ebar
